近日,我们高泽霞教授团队联合信息学院龚静教授团队在Nucleic Acids Research期刊上发表题为“iFish: a comprehensive multi-omics database for fish genetics, transcriptional regulation, and epigenetic research”的研究论文。该团队开发的“iFish”综合性鱼类多组学数据库(https://gonglab.hzau.edu.cn/iFish/),不仅整合了基因组图谱、基因表达谱、表观遗传修饰及蛋白质组数据,还实现了遗传变异、转录因子(TFs)及非编码RNA(ncRNAs)的系统鉴定。它是迄今为止系统整合、分析和存储鱼类多组学数据的最全面平台,将成为鱼类遗传学、转录调控和表观遗传学研究的重要资源。

随着高通量测序技术的发展,鱼类多组学数据的爆发式增长,正重塑水产领域的基础研究与应用研究范式。然而,数据分布碎片化、多组学整合度低等问题仍制约着数据的广泛使用。
因此,研究团队系统收集并整合了88种鱼类(13377个样本、近40 T原始测序数据)的基因组、转录组、表观组和蛋白质组数据集,对数据进行了统一过滤、流程处理和分析(图1)。基于884个全基因组测序数据集,研究团队鉴定出1.37亿个单核苷酸多态性(SNP)和4552万个插入/缺失变异(InDel)。基于9797个RNA测序(RNA-seq)、293个单细胞RNA测序(single-cell RNA-seq)以及840个microRNA(miRNA)测序数据集,研究团队对多个物种不同组织中的187万个信使RNA(mRNA)、287873个长链非编码RNA(lncRNA)、197554个环状RNA(circRNA)和6068个miRNA进行了鉴定和定量分析。通过整合1563个表观组学测序数据集,iFish提供了全基因组范围的组蛋白修饰、染色质可及性、DNA甲基化及染色质互作图谱。此外,数据库还纳入了50个蛋白质组学项目,用于蛋白质表达谱分析。为方便基因分析,研究团队收集了88种鱼类的基因组信息,整理了270万条基因注释条目,鉴定了同源基因,注释了192107个转录因子(TF),构建了转录因子调控网络和基因共表达网络,并对各类RNA进行了跨物种保守性分析。
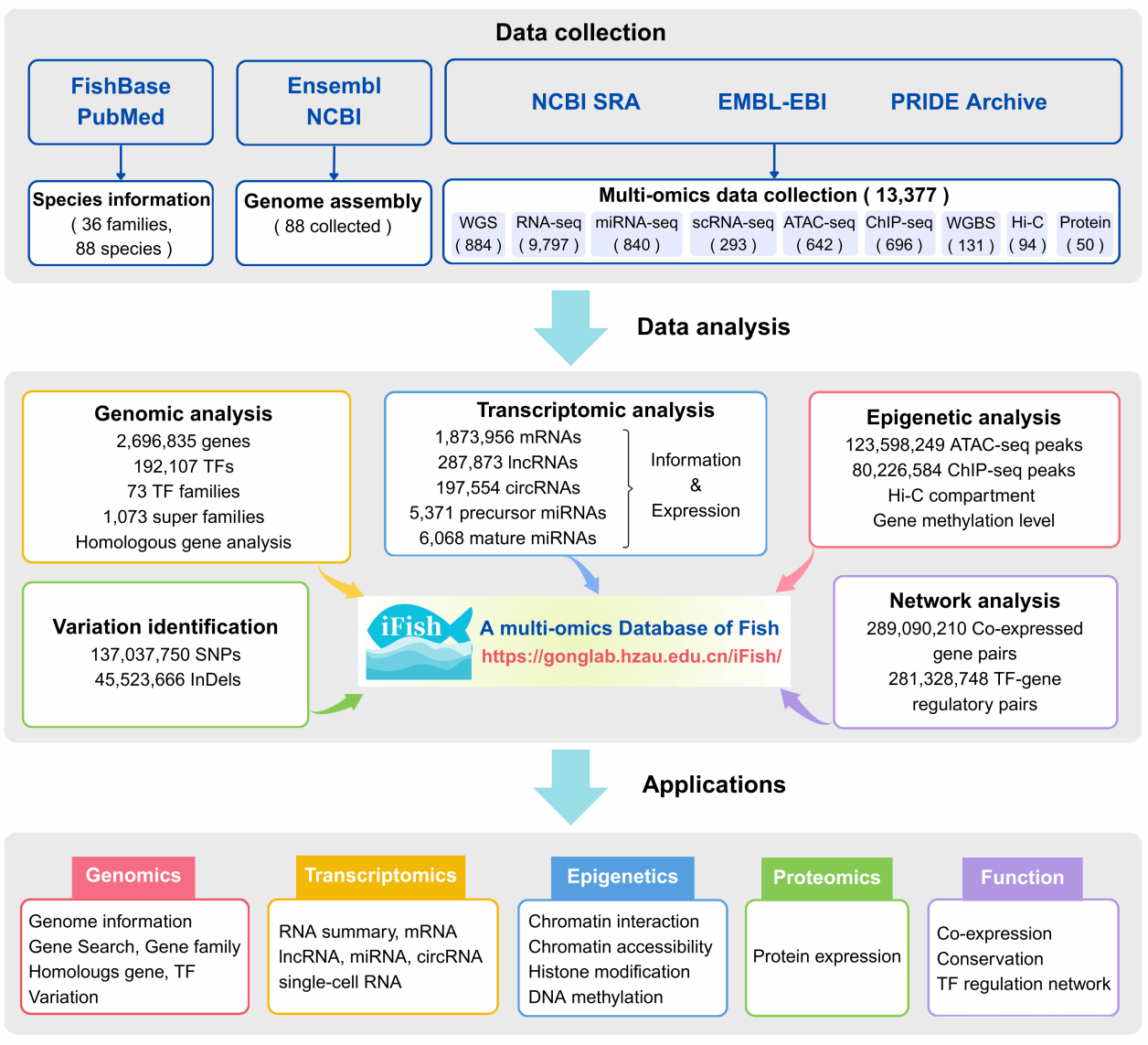
iFish数据库构建流程
iFish数据库不仅提供了基因注释、RNA表达谱分析、基因共表达网络和转录因子调控网络构建等核心生信分析工具,还配备交互式浏览、多维度可视化及便捷的数据下载功能,助力用户精准高效地开展查询与分析。iFish同时覆盖模式物种与经济物种,为全球鱼类科研工作者提供了“一站式”的数据检索、获取与深度挖掘服务,既为鱼类遗传学、转录调控机制、表观遗传学等基础研究提供便捷支撑,也为水产养殖遗传改良等产业实践提供数据参考。

iFish数据库核心功能
我校信息学院和我们联合培养研究生刘若妍和我们唐琴副研究员为该论文的共同第一作者,我们高泽霞教授和信息学院龚静教授为论文的共同通讯作者。该研究得到了国家自然科学基金项目、中央高校基本科研业务费专项资金、437ccm·必赢国际科技创新基金、国家大宗淡水鱼产业技术体系项目、湖北省重大科研项目等多个项目的资助。